
Eine Katalognummer für Organismen – eDNA, DNA-Barcoding und DNA-Metabarcoding
In einem vorherigen Beitrag haben wir das Thema Biodiversität grob umrissen und dabei die wesentlichen Aspekte beschrieben, die damit zu tun haben.
Jetzt wollen wir euch eine spannende Methode vorstellen, die in den letzten Jahren immer wichtiger geworden ist. Mit ihr kann man mit nur einer einzigen Probe einen Einblick in ein ganzes Ökosystem bekommen! Klingt zu cool, um wahr zu sein? Lest selbst:
Das Ordnen der Natur
Lebewesen anhand ähnlicher Merkmale zu ordnen und zu beschreiben ist einer der ältesten Zweige der Biologie und nennt sich Taxonomie. Ein Taxon (Plural: Taxa) beinhaltet dabei Lebewesen, die sich in einem oder mehreren Merkmalen ähnlich sind und bezüglich dessen als Einheit betrachtet werden können (z.B. alle Arthropoden, also Gliederfüßer wie Insekten, Spinnentiere, Krebstiere, oder alle Arten der Gattung Homo, wie der moderne Mensch Homo sapiens, aber auch der Neandertaler Homo neaderthalensis, et cetera). Das heutzutage verwendete taxonomische System zur eindeutigen Klassifikation von Lebewesen geht auf Carl von Linné zurück und weist jeder Art zwei Namen zu: Den Gattungsnamen (“Nachname”) und den Art- bzw: Speziesnamen. Z.B.: Triticum spelta (Dinkel), Cypripedium calceolus (Gelber Frauenschuh), Felis catus (Hauskatze) usw.
 Abb. 1: Carl von Linné (1707 – 1778) begründete das moderne taxonomische System zur eindeutigen Benennung der Arten. (http://mellifera.ch/cms/index.php/news/carl-von-linne-apis-mellifera)
Abb. 1: Carl von Linné (1707 – 1778) begründete das moderne taxonomische System zur eindeutigen Benennung der Arten. (http://mellifera.ch/cms/index.php/news/carl-von-linne-apis-mellifera)
Traditionellerweise wurden Arten seit hunderten von Jahren anhand äußerer Merkmale, bei Mikroorganismen auch unter dem Mikroskop und durch Vergleichen bestimmt bzw. klassifiziert und beschrieben. Das ist ein relativ komplizierter und zeitaufwendiger Prozess, der viel Fachwissen voraussetzt (aber durchaus nach wie vor seine Berechtigung hat und haben wird). Im Zuge der technischen Entwicklungen sind DNA-Analysen jedoch immer günstiger, schneller und genauer geworden. Sie spielen deshalb auch in der Erfassung und Messung von Biodiversität eine zunehmend wichtigere Rolle. Sehen wir uns das doch etwas genauer an.
Biodiversität erfassen – wozu überhaupt?
Menschengemachte Einflüsse verursachen nie dagewesene Veränderungen bezüglich des Verlustes an Biodiversität und damit auch der Ökosystemfunktionen. Wir brauchen deshalb entsprechende Werkzeuge, um Biodiversität zu überwachen und dadurch Informationen für konkrete Vorhaben wie Landnutzung, Renaturierung oder Naturschutz zu erhalten. Um den Zustand eines Ökosystems zu überwachen bzw. zu regeln, ist es unerlässlich, eine Art Inventar der dort vorkommenden Organismen und Biodiversitäts- Muster/Trends zu haben. Dadurch können folglich auch Renaturierungsmaßnahmen oder sonstige Veränderungen besser beurteilt werden.
Das Problem ist nur, dass die Bestimmung / Erfassung “von Hand” relativ langwierig und aufwändig ist. Zeitgleich steigt der Umfang an Proben kontinuierlich an, wohingegen Leute mit taxonomischer Expertise immer weniger werden. Und dann gibt es noch so viele Arten …
 Abb. 2: Der Stammbaum des Lebens mit seinen zahlreichen Arten. (Leonard Eisenberg via https://evogeneao.s3.amazonaws.com/images/tree_of_life/tree-of-life_2000.png bzw. https://www.evogeneao.com/de)
Abb. 2: Der Stammbaum des Lebens mit seinen zahlreichen Arten. (Leonard Eisenberg via https://evogeneao.s3.amazonaws.com/images/tree_of_life/tree-of-life_2000.png bzw. https://www.evogeneao.com/de)
Ein weiterer Nachteil könnte ebenfalls sein, dass man sich nur auf ein paar wenige bekannte oder charismatische Arten (“Wildbiene”, Pandabär) konzentriert und unbekannte, kleine oder unsichtbare Arten (z.B. Mikroben) gar nicht berücksichtigt werden. Außerdem erfordern einige Bestimmungsmethoden das Einfangen oder Töten von Individuen. Genau hier kommen die DNA-basierten Methoden der Biodiversitätserfassung ins Spiel.
Die molekularen Methoden der Biodiversitätserfassung – eDNA, DNA Barcoding und DNA Metabarcoding
Der große Vorteil dieser Werkzeuge ist, dass sie die oben genannten Probleme weitestgehend umgehen können (Insektenfallen sind dennoch oftmals unumgänglich). Sie haben sich zudem als schnelle und zuverlässige Methode zum Biodiversitätsmonitoring bereits bewährt.
Alles beginnt mit einer Umweltprobe (Wasser, Boden, Luft, Mageninhalt, Faeces, Biomasse et cetera), die “im Feld” genommen wird. Die Idee dahinter ist, dass jedes Lebewesen DNA enthält und dementsprechend auch DNA-Spuren in der Probe hinterlässt. Passenderweise steht eDNA somit auch für “environmental DNA”, also Umwelt-DNA. Danach wird im Labor zuerst die Gesamt-DNA aus der Probe extrahiert (also sämtliche DNA, die darin enthalten ist). Mithilfe der Polymerasekettenreaktion (PCR, auch schon lange vor Corona ein Standardwerkzeug in jedem molekularbiologischen Labor) wird dann ein bestimmter Teil der DNA vervielfältigt (meistens wird hierfür das cox1 Gen der Mitochondrien bei Tieren, Teile der Plastiden-DNA bei Pflanzen und die ITS1-Region bei Pilzen verwendet). Dann folgt die Sequenzierung, d.h. die genaue Abfolge der einzelnen DNA-Bausteine (die Basen Adenin, Guanin, Thymin und Cytosin) dieses vervielfältigten Abschnitts wird ermittelt. Danach erfolgt der Abgleich mit bekannten Sequenzen. Je nach Spezies unterscheiden sich die ausgelesenen DNA-Sequenzen mehr oder weniger stark und können mit Vergleichssequenzen in Datenbanken abgeglichen werden.
Falls eine Art schon aus früheren Studien des Meta-Barcodings bekannt ist, kann diese der entsprechenden Sequenz zugeordnet werden. Eine Zuordnung kann auch noch Jahre später erfolgen, wenn die Sequenz gespeichert wird. “Passt” eine bekannte Sequenz also zu einem DNA-Abschnitt aus der Probe, ist die Art nachgewiesen.
Abbildung 3 zeigt eine symbolhafte Beschreibung der DNA Metabarcoding Methode bei der Bestimmung von Insekten. Das grundsätzliche Verfahren ist aber für alle Umweltproben, die mit eDNA zu tun haben, dasselbe.
 Abb.3: Von der Umweltprobe zur individuellen DNA einzelner Arten. (https://www.waldwissen.net/de/lebensraum-wald/naturschutz/monitoring/metabarcoding)
Abb.3: Von der Umweltprobe zur individuellen DNA einzelner Arten. (https://www.waldwissen.net/de/lebensraum-wald/naturschutz/monitoring/metabarcoding)
Alleine schon die ermittelten DNA-Sequenzen selbst, also ohne Vergleichssequenzen, liefern Hinweise auf die Artenvielfalt und ein Maß der Biodiversität. Denn: Proben, deren Sequenzen einen Unterschied von über 3 % aufweisen, werden üblicherweise als unterschiedliche Arten gewertet. Kommt es also nicht primär darauf an, die einzelnen Arten namentlich anzusprechen, sind diese Daten für ökologische Fragestellungen sehr wertvoll. Man kann sich das dann wie so eine Art Barcode oder Inventarnummer vorstellen.
DNA Barcoding identifiziert somit bestimmte, einzelne Arten, deren DNA Sequenz bereits bekannt ist, aus einer Umweltprobe (Abbildung 4).
 Abb. 4: Beim DNA-Barcoding können Arten anhand ihrer bereits bekannten DNA-Sequenz aus einer Umweltprobe identifiziert werden. (https://www.lfu.bayern.de/analytik_stoffe/innovative_analysen/dna_verfahren/dna_barcoding/index.htm#:~:text=Eine%20DNA%2DBarcode%2DSequenz%20setzt,der%20Begriff%20DNA%2DBarcoding%20gew%C3%A4hlt.)
Abb. 4: Beim DNA-Barcoding können Arten anhand ihrer bereits bekannten DNA-Sequenz aus einer Umweltprobe identifiziert werden. (https://www.lfu.bayern.de/analytik_stoffe/innovative_analysen/dna_verfahren/dna_barcoding/index.htm#:~:text=Eine%20DNA%2DBarcode%2DSequenz%20setzt,der%20Begriff%20DNA%2DBarcoding%20gew%C3%A4hlt.)
Beim DNA Metabarcoding werden dabei mehrere Arten bzw. Taxa gleichzeitig identifiziert.Abbildung 5 verdeutlicht die Unterschiede.
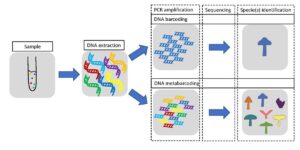 Abb. 5: DNA Barcoding identifiziert einzelne Arten, DNA Metabarcoding identifiziert mehrere Arten und ist somit quasi eine “erweiterte” Form. (https://en.wikipedia.org/wiki/File:DNA_(meta)barcoding_differences.pdf)
Abb. 5: DNA Barcoding identifiziert einzelne Arten, DNA Metabarcoding identifiziert mehrere Arten und ist somit quasi eine “erweiterte” Form. (https://en.wikipedia.org/wiki/File:DNA_(meta)barcoding_differences.pdf)
DNA Barcoding bzw. Metabarcoding stellt somit zusätzlich zur klassischen Taxonomie ein nützliches Werkzeug zur Erfassung der Biodiversität dar, denn es hilft uns zusätzlich, Biodiversität greifbarer und weniger abstrakt zu machen. Dies ist von unschätzbarem Wert und zwar auf mehrere Weisen. Zum einen ist es ein zusätzliches Werkzeug, um den “Ist-Zustand” zu bewerten. Zum anderen, und das ist wahrscheinlich noch wichtiger, eignet es sich hervorragend zum kontinuierlichen Monitoring und damit zur Bewertung von z.B. Renaturierungsmaßnahmen. Denn es können ja auch Organismen nachgewiesen werden, die zwar aktuell gerade nicht in der Probe vorhanden sind, aber ihre Spuren in Form von DNA hinterlassen haben. Wir gewinnen durch diese Methoden der Molekularbiologie also einen umfassenderen Blick auf ein Ökosystem und gerade jetzt können wir jede Hilfe gut gebrauchen.
Quelle Titelbild: Pixabay/Mylene2401
Literatur
Boenigk, Jens ; Wodniok, Sabina: Biodiversität und Erdgeschichte. Berlin Heidelberg New York: Springer-Verlag, 2014. S. 164-165.
http://mellifera.ch/cms/index.php/news/carl-von-linne-apis-mellifera
https://besjournals.onlinelibrary.wiley.com/doi/full/10.1111/2041-210X.12574
https://en.wikipedia.org/wiki/Metabarcoding
https://onlinelibrary.wiley.com/doi/full/10.1111/mec.14350
https://www.nature.com/articles/nature11148?report=reader
https://www.science.org/doi/abs/10.1126/science.1251156
https://www.sciencedirect.com/science/article/abs/pii/S1877343512000085
https://www.spektrum.de/lexikon/biologie/linne-carl-von/39482
https://www.waldwissen.net/de/lebensraum-wald/naturschutz/monitoring/metabarcoding
Sadava, David ; Hillis, David M. ; Heller, H. Craig ; Hacker, Sally D.: Purves Biologie. Berlin Heidelberg New York: Springer-Verlag, 2019. S. 663-668
Titelbild: https://www.agri-pulse.com/ext/resources/2021/05/11/DNA_Barcoding_836x627.png?1620771493
Alle Internetseiten zuletzt abgerufen am 18.12.2022
Getagged mit:biodiversität, biotechnologie, cbd, cop15, DNA Barcoding, eDNA, umwelt, umweltschutz, weltbiodiversitätskonferenz
Comments & Reviews Antworten abbrechen
Diese Website verwendet Akismet, um Spam zu reduzieren. Erfahre mehr darüber, wie deine Kommentardaten verarbeitet werden.

